Pemodelan Pohon Klasifikasi Menggunakan Rpart


Pada tulisan sebelumnya, kita sudah membahas tentang teori dan konsep di balik pemodelan pohon klasifikasi. Pembahasan meliputi konsep, terminologi dan komponen pada pohon klasifikasi. Selain itu juga dibahas tentang algoritma dasar yang dilengkapi ilustrasi bagaimana pohon klasifikasi tersebut dibangun. Adapun fokus pada tulisan ini adalah bagaimana penerapan model pohon klasifikasi tersebut menggunakan bahasa pemrograman R.
Baca: Pohon Klasifikasi: Teori dan Konsep Dasar
Di dalam bahasa R, terdapat beberapa pustaka yang dapat digunakan untuk membuat model pohon klasifikasi, salah satunya adalah rpart. Pada tutorial ini, kita akan menggunakan pustaka rpart tersebut untuk membangun model pohon klasifikasi. Data yang akan digunakan adalah wine quality binary dataset.
Penyiapan Data
Dataset yang digunakan memiliki format csv sehingga kita dapat membacanya menggunakan fungsi read.csv. Peubah respon atau kelas pada dataset tersimpan pada kolom quality dengan nilai LOW atau HIGH. Sementara kolom lainnya seluruhnya bertipe numerik.
R
data <- read.csv("wine-quality-binary.csv")
str(data)# OUTPUT 'data.frame': 1143 obs. of 13 variables: $ id : int 1 2 3 4 5 6 7 8 9 10 ... $ fixed.acidity : num 7.4 7.8 7.8 11.2 7.4 7.4 7.9 7.3 7.8 6.7 ... $ volatile.acidity : num 0.7 0.88 0.76 0.28 0.7 0.66 0.6 0.65 0.58 0.58 ... $ citric.acid : num 0 0 0.04 0.56 0 0 0.06 0 0.02 0.08 ... $ residual.sugar : num 1.9 2.6 2.3 1.9 1.9 1.8 1.6 1.2 2 1.8 ... $ chlorides : num 0.076 0.098 0.092 0.075 0.076 0.075 0.069 0.065 0.073 0.097 ... $ free.sulfur.dioxide : num 11 25 15 17 11 13 15 15 9 15 ... $ total.sulfur.dioxide: num 34 67 54 60 34 40 59 21 18 65 ... $ density : num 0.998 0.997 0.997 0.998 0.998 ... $ pH : num 3.51 3.2 3.26 3.16 3.51 3.51 3.3 3.39 3.36 3.28 ... $ sulphates : num 0.56 0.68 0.65 0.58 0.56 0.56 0.46 0.47 0.57 0.54 ... $ alcohol : num 9.4 9.8 9.8 9.8 9.4 9.4 9.4 10 9.5 9.2 ... $ quality : chr "LOW" "LOW" "LOW" "HIGH" ...
Dari struktur di atas dapat pula kita lihat terdapat kolom id. Jika diperhatikan, kolom ini hanya berisi urutan data dari 1, 2… sampai 1143 sesuai banyaknya data. Kolom ini artinya bukanlah peubah yang berkaitan dengan kelas data sehingga bisa kita hapus saja. Kita juga sebaiknya merubah tipe dari peubah respon menjadi factor. Hal ini nantinya dapat mempermudah pengukuran pada proses-proses selanjutnya.
Pada kode di bawah ini, kita juga menampilkan banyaknya data serta komposisinya menurut peubah quality. Dari total 1143 observasi, terdapat 621 yang masuk sebagai kelas "HIGH" dan 522 sebagai kelas "LOW". Berdasarkan proporsinya kedua kelas relatif seimbang yaitu dengan komposisi sekitar 54% data dengan kelas "HIGH" berbanding 46% data dengan kelas "LOW".
R
# menghapus kolom `id` data$id <- NULL # mengubah tipe kolom `quality` menjadi factor data$quality <- as.factor(data$quality) # melihat jumlah dan komposisi masing-masing kategori quality <- data.frame(table(data$quality)) quality$Prop <- round(quality$Freq/sum(quality$Freq), 3) print(quality)
# OUTPUT Var1 Freq Prop 1 HIGH 621 0.543 2 LOW 522 0.457
Pembagian Data Latih dan Data Uji
Membagi data menjadi data latih dan data uji merupakan tahapan penting dalam pemodelan machine learning termasuk pohon klasifikasi. Pembagian ini diperlukan agar model dapat diuji menggunakan data yang belum pernah dilihat selama proses pelatihan. Pada saat pelatihan model, data yang digunakan hanya data latih saja. Adapun data uji nantinya digunakan untuk mengukur performa model. Hal ini penting agar pengukuran performa lebih “fair” karena menggunakan data baru yang belum pernah digunakan untuk proses pelatihan.
Pembagian data pada bahasa R dapat dilakukan menggunakan fungsi createDataPartition dari pustaka caret. Proporsi banyaknya data yang nantinya digunakan untuk melatih model dan berapa banyak untuk pengujian dapat ditentukan dengan mengatur parameter p. Pada contoh ini, kita akan menggunakan 75% dataset sebagai data latih dan 25% sisanya menjadi data uji.
R
# Membagi data sebagai data Latih dan data uji
library(caret)
set.seed(123) # Untuk menghasilkan nilai acak yang dapat direproduksi (pesudo-random)
# membagi data secara acak
# dengan menghasilkan indeks data latih dan data uji
# data latih = 75%, data uji = 25%
trainIndex <- createDataPartition(data$quality, p = 0.75, list = FALSE, times = 1)
# Buat data latih dan data uji berdasarkan indeks yang dihasilkan
data.train <- data[trainIndex, ]
data.test <- data[-trainIndex, ]
# melihat komposisi setiap kelas pada data train dan test
cbind("train" = table(data.train$quality), "test" = table(data.test$quality))# OUTPUT
train test
HIGH 466 155
LOW 392 130Model Pohon Klasifikasi
Membuat Model
Untuk membuat model pohon klasifikasi di R dengan pustaka rpart, kita dapat memanggil fungsi rpart. Parameter pertama yaitu formula. Pada contoh kode di bawah ini quality ~ . menyatakan bahwa peubah respon yang akan diprediksi adalah quality sementara peubah lainnya yang terdapat pada data digunakan sebagai prediktor. Data yang digunakan dalam proses pelatihan adalah data.train sementara method adalah "class" yang menunjukkan pemodelan klasifikasi.
Output yang dihasilkan adalah sekumpulan aturan yang menunjukkan bagaimana data dipartisi ke dalam simpul-simpul sesuai aturan partisi yang terbentuk. Sebagai contoh pada simpul akar (simpul 1) data dipartisi menggunakan variabel alcohol dengan batasan nilai alcohol>=9.85. Setiap data yang memenuhi kriteria ini akan dipartisi (simpul 2) dan dipisahkan dengan data yang tidak memenuhi kriteria (simpul 3). Setiap simpul baru yang terbentuk diproses kembali dengan cara yang sama dan menghasilkan simpul baru ataupun menjadi simpul akhir sesuai kondisi yang ditentukan.
Representasi pohon klasifikasi dapat dibuat dalam bentuk diagram pohon. Visualisasi ini akan lebih memudahkan kita untuk memahami bagaimana proses partisi dilakukan serta variabel dan nilai apa yang digunakan dalam proses tersebut.
R
library(rpart) library(rpart.plot) model.cart <- rpart(quality ~ ., data=data.train, method = "class") model.cart # visualisasi pohon klasifikasi rpart.plot(model.cart)
# OUTPUT
n= 858
node), split, n, loss, yval, (yprob)
* denotes terminal node
1) root 858 392 HIGH (0.5431235 0.4568765)
2) alcohol>=9.85 528 157 HIGH (0.7026515 0.2973485)
4) sulphates>=0.585 368 75 HIGH (0.7961957 0.2038043)
8) total.sulfur.dioxide< 85.5 347 62 HIGH (0.8213256 0.1786744) *
9) total.sulfur.dioxide>=85.5 21 8 LOW (0.3809524 0.6190476)
18) residual.sugar< 2.25 10 2 HIGH (0.8000000 0.2000000) *
19) residual.sugar>=2.25 11 0 LOW (0.0000000 1.0000000) *
5) sulphates< 0.585 160 78 LOW (0.4875000 0.5125000)
10) alcohol>=11.45 43 8 HIGH (0.8139535 0.1860465) *
11) alcohol< 11.45 117 43 LOW (0.3675214 0.6324786)
22) volatile.acidity< 0.585 54 26 HIGH (0.5185185 0.4814815)
44) sulphates>=0.48 47 19 HIGH (0.5957447 0.4042553)
88) density< 0.996425 31 8 HIGH (0.7419355 0.2580645) *
89) density>=0.996425 16 5 LOW (0.3125000 0.6875000) *
45) sulphates< 0.48 7 0 LOW (0.0000000 1.0000000) *
23) volatile.acidity>=0.585 63 15 LOW (0.2380952 0.7619048) *
3) alcohol< 9.85 330 95 LOW (0.2878788 0.7121212)
6) sulphates>=0.585 179 74 LOW (0.4134078 0.5865922)
12) volatile.acidity< 0.405 38 10 HIGH (0.7368421 0.2631579) *
13) volatile.acidity>=0.405 141 46 LOW (0.3262411 0.6737589)
26) total.sulfur.dioxide< 28.5 41 16 HIGH (0.6097561 0.3902439)
52) volatile.acidity< 0.555 18 2 HIGH (0.8888889 0.1111111) *
53) volatile.acidity>=0.555 23 9 LOW (0.3913043 0.6086957) *
27) total.sulfur.dioxide>=28.5 100 21 LOW (0.2100000 0.7900000) *
7) sulphates< 0.585 151 21 LOW (0.1390728 0.8609272) *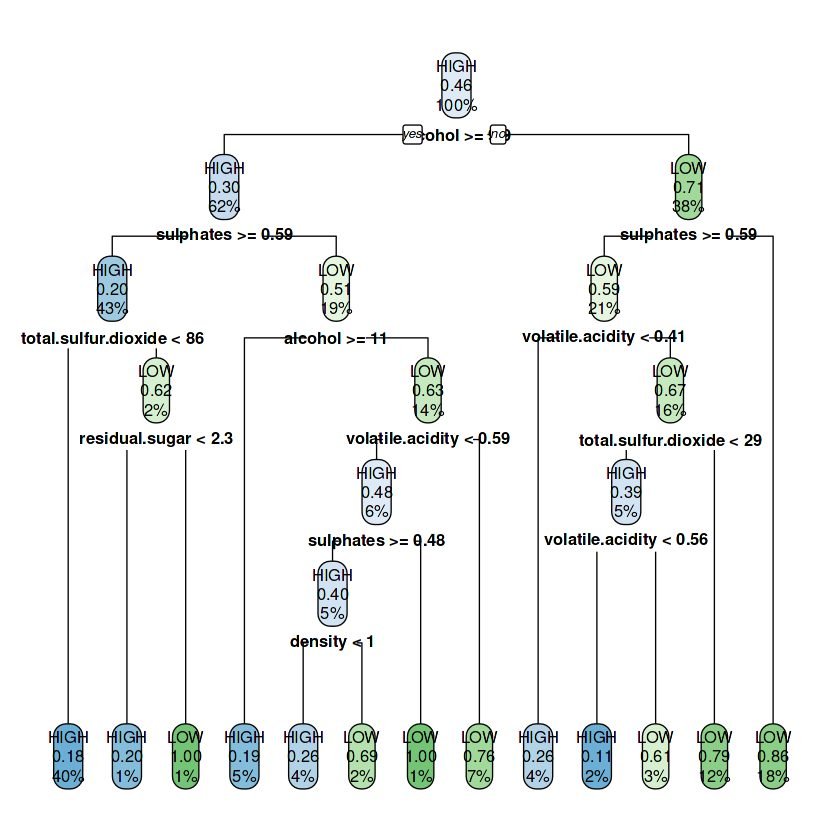

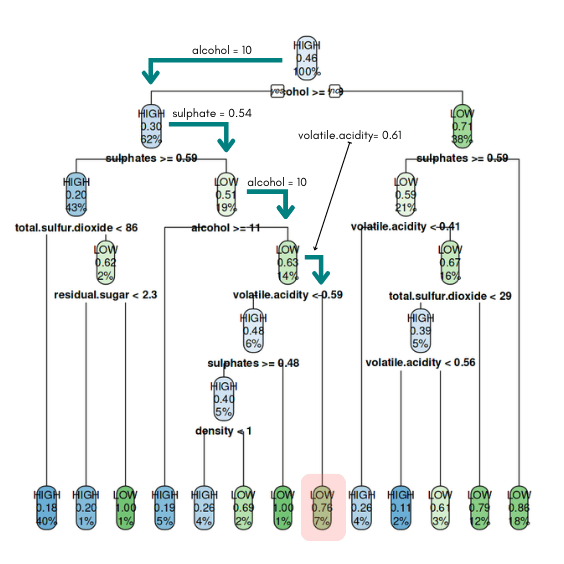
Dari pohon tersebut misalkan terdapat data wine baru dengan komposisi sebagai berikut:
fixed.acidity: 11.2volatile.acidity: 0.61citric.acid: 0.08residual.sugar: 1.9chlorides: 0.075free.sulfur.dioxide: 15total.sulfur.dioxide: 94density: 0.998pH: 3.39sulphates: 0.54alcohol: 10
Maka wine tersebut akan diprediksi sebagai LOW. Adapun alurnya dapat dilihat pada gambar di bawah ini. Pada tahap pertama, akan dicek apakah nilai variabel alcohol >= 9.85 karena nilainya benar maka data ini akan masuk ke dalam simpul di sebelah kiri (yes). Proses selanjutnya adalah mengecek apakah nilai sulphate >= 0.59, dimana untuk data ini bernilai 0.54 sehingga akan masuk ke simpul bagian kanan (no). Proses berlanjut sampai pada bagian akhir dimana data masuk pada simpul daun dan diprediksi sebagai kelas "LOW".

Prediksi Data Uji
Pada tahap awal, dataset sudah kita bagi menjadi data latih dan data uji. Pohon klasifikasi dilatih menggunakan data latih saja. Setelah model dibuat maka kita perlu menguji seberapa baik performa model dalam memprediksi data baru. Di sinilah peran dari data uji dimana data ini sudah memiliki label kelas LOW atau HIGH. Kelas yang didapatkan dari hasil prediksi nantinya akan dibandingkan dengan label kelas sebenarnya dari data uji ini.
Prediksi data baru dilakukan menggunakan fungsi predict. Di dalam fungsi predict kita perlu menentukan parameter model yang akan digunakan untuk prediksi serta data yang akan diprediksi. Adapun untuk parameter type, dapat menggunakan class karena peubah respon sudah dalam tipe data factor. Dengan mengatur menjadi class maka output hasil prediksi langsung berupa kelas sesuai data pelatihan (pada contoh ini yaitu HIGH atau LOW).
R
# Memprediksi data uji
pred.test <- predict(model.cart, newdata = data.test, type="class")
# Melihat prediksi pada 10 data.test yang pertama
head(pred.test, 10)
# Membuat Confusion Matrix
confusionMatrix(data = pred.test,
reference = data.test$quality)# OUTPUT
# Contoh hasil prediksi 10 data test pertama
1 3 4 7 8 9 10 17 20 23
LOW LOW LOW LOW LOW LOW LOW LOW HIGH LOW
Levels: HIGH LOW
# Confusion Matrix and Statistics
Reference
Prediction HIGH LOW
HIGH 121 35
LOW 34 95
Accuracy : 0.7579
95% CI : (0.7039, 0.8065)
No Information Rate : 0.5439
P-Value [Acc > NIR] : 6.256e-14
Kappa : 0.5117
Mcnemar's Test P-Value : 1
Sensitivity : 0.7806
Specificity : 0.7308
Pos Pred Value : 0.7756
Neg Pred Value : 0.7364
Prevalence : 0.5439
Detection Rate : 0.4246
Detection Prevalence : 0.5474
Balanced Accuracy : 0.7557
'Positive' Class : HIGH
Hasil prediksi dapat dibandingkan dengan kelas yang sebenarnya menggunakan confusion matrix. Di dalam bahasa R, kita dapat menggunakan fungsi confusionMatrix dari pustaka caret untuk membuat confusion matrix. Selain itu, fungsi ini juga memberikan informasi beberapa ukuran performa model seperti akurasi, sensitivitas dan spesifisitas. Terdapat beberapa parameter yang dapat diatur, dua yang utama adalah data dan reference. Parameter data berisi label kelas hasil prediksi, sementara parameter reference berisi kelas sebenarnya dari data uji.
Berdasarkan hasil di atas diperoleh nilai akurasi model sebesar 0.7579. Nilai ini menunjukkan bahwa model mampu memprediksi secara tepat 75.79% kelas dari data uji. Lebih detail, dari 155 data uji yang merupakan kelas HIGH, terdapat 121 yang diprediksi benar, sementara 34 lainnya salah prediksi atau memiliki nilai sensitivity sebesar 0.7806. Sebaliknya dari 120 data uji yang merupakan kelas LOW, 95 diprediksi secara tepat dan 35 lainnya salah atau memiliki nilai specificity sebesar 0.7308. Selain itu terdapat beberapa ukuran lainnya yang juga ditampilkan dari output fungsi confusionMatrix.
Pengaturan Hiperparameter
Hiperparameter Pohon Klasifikasi
Saat pembentukan pohon klasifikasi, terdapat beberapa hiperparameter yang dapat ditentukan. Pada model sebelumnya kita hanya menagtur hiperparameter tersebut dengan nilai defaultnya saja. Berikut beberapa hiperparameter untuk pohon klasifikasi menggunakan melalui fungsi rpart.control:
minbucket(default=minsplit/3): Jumlah minimum observasi yang harus ada di sebuah simpul agar pemisahan dapat dilakukan.minsplit(default=20): Jumlah minimum observasi di setiap simpul akhir (daun). Jika hanya salah satu dariminbucketatauminsplityang diatur, maka kode akan mengaturminsplit = minbucket*3atauminbucketmenjadiminsplit/3, sesuai kebutuhan.*cp(default=0.01): Parameter kompleksitas, peran utama dari parameter ini adalah untuk menghemat waktu komputasi dengan memangkas pemisahan yang jelas-jelas tidak layak. Pada dasarnya, mengatur bahwa setiap pemisahan yang tidak meningkatkan kecocokan sebanyakcpkemungkinan akan dipangkas dalam validasi silang, dan oleh karena itu program tidak perlu memproses lebih lanjut.xval(default=10): Jumlah validasi silang.maxdepth(default=30): Mengatur kedalaman maksimum dari setiap simpul pohon final, dengan node akar dihitung sebagai kedalaman 0.
Selain hiperparameter tersebut terdapat beberapa hiperparameter lainnya. Selengkapnya dapat dilihat pada dokumentasi rpart.control.
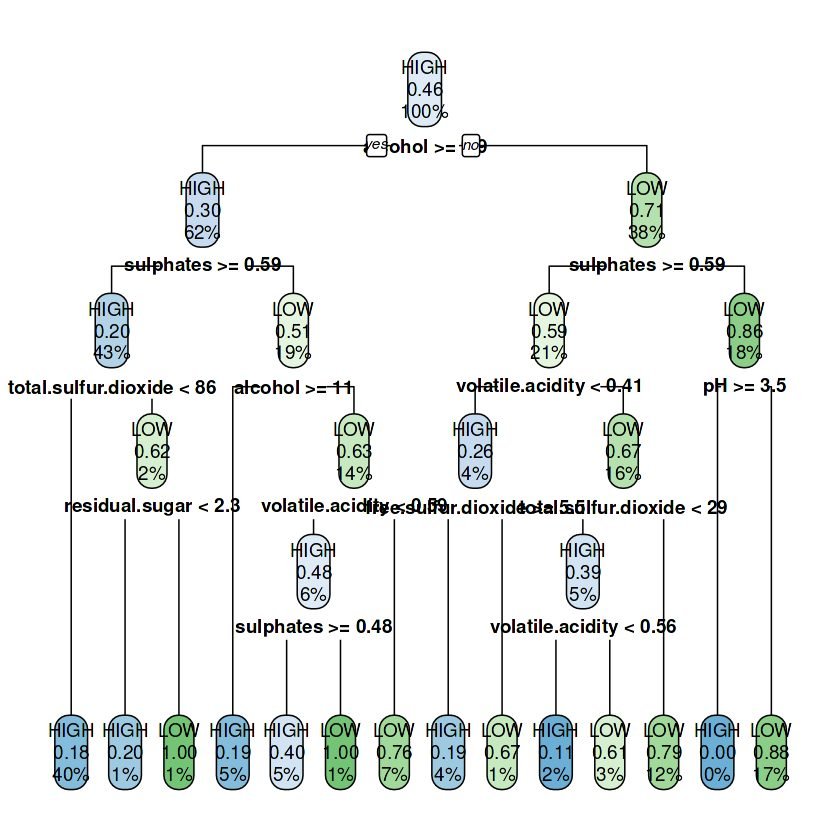
Pada contoh kode berikut ini kita akan membuat model pohon klasifikasi serta mengatur beberapa hiperparameter (minsplit, cp dan maxdepth) dengan nilai tertentu menggunakan fungsi rpart.control. Selanjutnya nilai-nilai tersebut digunakan pada fungsi rpart sebagai argumen untuk parameter control.
R
# rpart.control : mengatur nilai parameter model (lihat : ?rpart.control) control = rpart.control(minsplit = 5, cp = 0.005, maxdepth = 5) model.cart.2 <- rpart(quality ~ ., data=data.train, method = "class", control = control) model.cart.2 rpart.plot(model.cart.2)
# OUTPUT
n= 858
node), split, n, loss, yval, (yprob)
* denotes terminal node
1) root 858 392 HIGH (0.5431235 0.4568765)
2) alcohol>=9.85 528 157 HIGH (0.7026515 0.2973485)
4) sulphates>=0.585 368 75 HIGH (0.7961957 0.2038043)
8) total.sulfur.dioxide< 85.5 347 62 HIGH (0.8213256 0.1786744) *
9) total.sulfur.dioxide>=85.5 21 8 LOW (0.3809524 0.6190476)
18) residual.sugar< 2.25 10 2 HIGH (0.8000000 0.2000000) *
19) residual.sugar>=2.25 11 0 LOW (0.0000000 1.0000000) *
5) sulphates< 0.585 160 78 LOW (0.4875000 0.5125000)
10) alcohol>=11.45 43 8 HIGH (0.8139535 0.1860465) *
11) alcohol< 11.45 117 43 LOW (0.3675214 0.6324786)
22) volatile.acidity< 0.585 54 26 HIGH (0.5185185 0.4814815)
44) sulphates>=0.48 47 19 HIGH (0.5957447 0.4042553) *
45) sulphates< 0.48 7 0 LOW (0.0000000 1.0000000) *
23) volatile.acidity>=0.585 63 15 LOW (0.2380952 0.7619048) *
3) alcohol< 9.85 330 95 LOW (0.2878788 0.7121212)
6) sulphates>=0.585 179 74 LOW (0.4134078 0.5865922)
12) volatile.acidity< 0.405 38 10 HIGH (0.7368421 0.2631579)
24) free.sulfur.dioxide>=5.5 32 6 HIGH (0.8125000 0.1875000) *
25) free.sulfur.dioxide< 5.5 6 2 LOW (0.3333333 0.6666667) *
13) volatile.acidity>=0.405 141 46 LOW (0.3262411 0.6737589)
26) total.sulfur.dioxide< 28.5 41 16 HIGH (0.6097561 0.3902439)
52) volatile.acidity< 0.555 18 2 HIGH (0.8888889 0.1111111) *
53) volatile.acidity>=0.555 23 9 LOW (0.3913043 0.6086957) *
27) total.sulfur.dioxide>=28.5 100 21 LOW (0.2100000 0.7900000) *
7) sulphates< 0.585 151 21 LOW (0.1390728 0.8609272)
14) pH>=3.545 3 0 HIGH (1.0000000 0.0000000) *
15) pH< 3.545 148 18 LOW (0.1216216 0.8783784) *
'Positive' Class : HIGH

Prediksi Data Uji
Sama seperti model sebelumnya, kita perlu melakukan prediksi pada data uji untuk mengukur performa model. Berdasarkan hasil berikut, diperoleh nilai akurasi model sebesar 0.7719 atau model mampu memprediksi secara tepat 77.19% kelas dari data uji. Nilai ini lebih tinggi dibandingkan yang diperoleh pada model sebelumnya yaitu sebesar 0.7579. Begitu pula dengan nilai sensitivitas model yang mencapai 0.8000 dan spesifisitas sebesar 0.7385 keduanya memberikan hasil yang lebih tinggi dibandingkan model sebelumnya.
R
# Memprediksi data uji
pred.test.2 <- predict(model.cart.2, newdata = data.test, type="class")
# Melihat prediksi pada 10 data.test yang pertama
head(pred.test.2, 10)
# Membuat Confusion Matrix
confusionMatrix(data = pred.test.2,
reference = data.test$quality)# OUTPUT
# Prediksi 10 data test pertama
1 3 4 7 8 9 10 17 20 23
LOW LOW LOW LOW LOW LOW LOW LOW HIGH LOW
Levels: HIGH LOW
# Confusion Matrix and Statistics
Reference
Prediction HIGH LOW
HIGH 124 34
LOW 31 96
Accuracy : 0.7719
95% CI : (0.7188, 0.8193)
No Information Rate : 0.5439
P-Value [Acc > NIR] : 1.102e-15
Kappa : 0.5395
Mcnemar's Test P-Value : 0.8041
Sensitivity : 0.8000
Specificity : 0.7385
Pos Pred Value : 0.7848
Neg Pred Value : 0.7559
Prevalence : 0.5439
Detection Rate : 0.4351
Detection Prevalence : 0.5544
Balanced Accuracy : 0.7692
'Positive' Class : HIGH
Peubah Penting (Variable Importance)
Variable Importance adalah salah satu alat pada model berbasis pohon untuk mengetahui peubah prediktor mana yang memiliki kontribusi besar dalam memprediksi data. Secara lebih teknis, nilai-nilai dalam variable.importance dihasilkan dengan cara mengukur kontribusi setiap variabel prediktor dalam meningkatkan kualitas model. Nilai ini dihitung berdasarkan seberapa baik variabel-variabel tersebut dapat memisahkan atau mengklasifikasikan data pada setiap simpul dalam pohon. Semakin tinggi nilai variable.importance, semakin besar kontribusi variabel tersebut dalam membantu model membuat keputusan. Jika diperlukan pada model akhir, kita dapat menghapus variabel prediktor yang mungkin memiliki tingkat kepentingan yang sangat kecil. Penghapusan dapat mengurangi kompleksitas dari model tanpa banyak mengurangi performa model itu sendiri.
Dari hasil berikut, dapat dilihat bahwa variabel alcohol memiliki nilai variable.importance tertinggi atau dapat dikatakan bahwa variabel ini merupakan variabel paling penting dalam penentuan hasil prediksi model. Variable lainnya yang memiliki tingkat kepentingan tertinggi adalah sulphates dan total.sulfur.dioxide. Sementara itu, pH dan chlorides merupakan dua variabel dengan tingkat kepentingan paling kecil atau pengaruhnya sangat kecil dalam penentuan hasil prediksi.
R
# menampilkan nilai variable importance
print(model.cart.2$variable.importance)
# visualissi variable importace dengan dotchart
dotchart(sort(model.cart.2$variable.importance, decreasing = F),
col="darkgreen", bg="darkgreen")# OUTPUT
alcohol sulphates total.sulfur.dioxide
82.938043 42.078034 30.063096
volatile.acidity density free.sulfur.dioxide
29.781262 21.321031 13.070953
citric.acid fixed.acidity residual.sugar
10.080188 10.037989 9.782451
chlorides pH
8.270775 7.710209 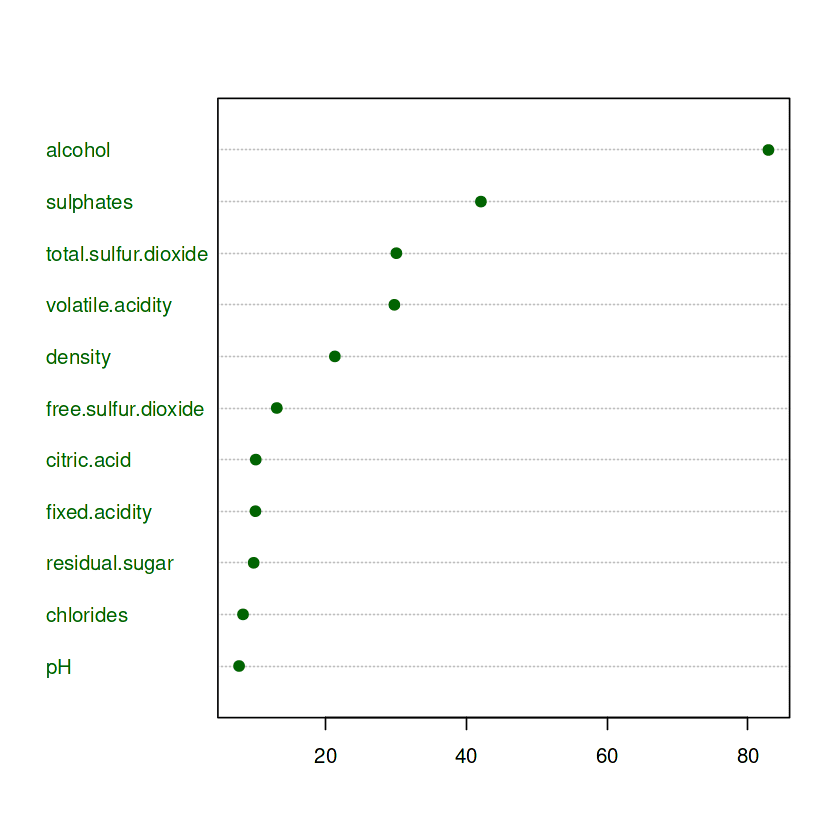

Ensemble Learning (Pohon Gabungan)
Pohon klasifikasi merupakan model sederhana dengan membangun 1 pohon keputusan saja. Penggunaan 1 pohon ini tentu saja sangat rentan terhadap perbedaan data saat melakukan pengacakan data latih dan data uji. Dengan pengacakan yang berbeda, besar kemungkinan akan membentuk sturktur pohon yang berbeda pula. Oleh karena itu dikembangkan berbagai model berbasis pohon gabungan. Keputusan tidak didasarkan dari 1 pohon saja, namun dari banyak pohon sekaligus. Dalam kasus pemodelan klasifikasi maka keputusan ditentukan dengan suara terbanyak (majority vote) berdasarkan hasil prediksi setiap pohon.
Model berbasis pohon gabungan yang banyak digunakan karena kemampuan prediksinya sangat baik diantaranya adalah Random Forest dan Gradient Boosting.
Berikut tutorial model pembelajaran mesin menggunakan Ensemble Learning menggunakan bahasa R:
Jika tertarik untuk mempelajarinya dalam bahasa python berikut tautannya: