Tuning Hyperparameter Model Random Forest dengan Bahasa R


Sekilas Random Forest
Random Forest adalah model ensemble berbasis pohon yang populer pada machine learning. Model ini diperkenalkan oleh Leo Breiman pada Tahun 2001. Random Forest dapat diterapkan pada pemodelan regresi maupun klasifikasi. Pada model random forest untuk regresi prediksi dihitung berdasarkan nilai rata-rata (averaging) dari output setiap decision tree. Sedangkan untuk model klasifikasi, prediksi ditentukan menggunakan suara terbanyak (majority vote). Contohnya, model Random Forest dengan 100 pohon dengan 72 pohon memprediksi data tertentu sebagai kelas A dan 28 pohon memprediksi masuk kelas B, maka data tersebut akan diprediksi sebagai kelas A.
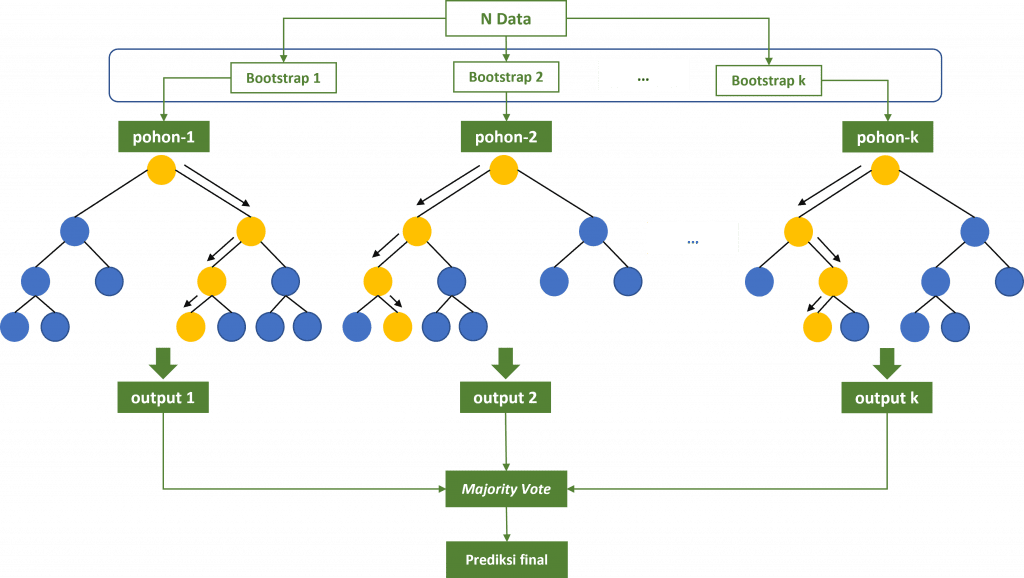

Tutorial ini membahas pembuatan model random forest menggunakan Bahasa R khususnya dengan paket caret dan randomForest.
[Terdapat paket lain yang powerful serta lebih terintegrasi yaitu tidymodels atau mlr3. Tutorial random forest dengan tidymodels dapat dilihat di sini Membangun Model Machine Learning dengan tidymodels R dan tutorial dengan mlr3 dapat dilihat di sini Membangun Model Random Forest dengan mlr3 di R]
Data
Data yang akan digunakan adalah dataset Sonar yang tersedia pada package mlbench. Dataset Sonar terdiri dari 208 sampel, di mana setiap sampel mewakili pola sonar yang dipantulkan oleh sebuah objek di bawah air. Setiap sampel memiliki 60 fitur numerik yang menyatakan kekuatan sinyal sonar pada frekuensi tertentu. Fitur-fitur ini direpresentasikan sebagai bilangan real antara 0,0 hingga 1,0. Tujuannya adalah untuk membedakan antara sinyal sonar yang dipantulkan oleh batuan (kelas “R”) dan sinyal sonar yang dipantulkan oleh tambang logam (kelas “M”).
Package caret dan randomForest digunakan untuk melatih model Random Forest serta melakukan tuning hyperparameter.
R
library(caret) library(mlbench) library(randomForest) # Load Dataset Sonar data(Sonar) summary(Sonar)
Output
V1 V2 V3 V4 V5 V6 V7
Min. :0.00150 Min. :0.00060 Min. :0.00150 Min. :0.00580 Min. :0.00670 Min. :0.01020 Min. :0.0033
1st Qu.:0.01335 1st Qu.:0.01645 1st Qu.:0.01895 1st Qu.:0.02438 1st Qu.:0.03805 1st Qu.:0.06703 1st Qu.:0.0809
Median :0.02280 Median :0.03080 Median :0.03430 Median :0.04405 Median :0.06250 Median :0.09215 Median :0.1070
Mean :0.02916 Mean :0.03844 Mean :0.04383 Mean :0.05389 Mean :0.07520 Mean :0.10457 Mean :0.1217
3rd Qu.:0.03555 3rd Qu.:0.04795 3rd Qu.:0.05795 3rd Qu.:0.06450 3rd Qu.:0.10028 3rd Qu.:0.13412 3rd Qu.:0.1540
Max. :0.13710 Max. :0.23390 Max. :0.30590 Max. :0.42640 Max. :0.40100 Max. :0.38230 Max. :0.3729
...
V50 V51 V52 V53 V54 V55 V56
Min. :0.00000 Min. :0.000000 Min. :0.000800 Min. :0.000500 Min. :0.001000 Min. :0.00060 Min. :0.000400
1st Qu.:0.01155 1st Qu.:0.008425 1st Qu.:0.007275 1st Qu.:0.005075 1st Qu.:0.005375 1st Qu.:0.00415 1st Qu.:0.004400
Median :0.01790 Median :0.013900 Median :0.011400 Median :0.009550 Median :0.009300 Median :0.00750 Median :0.006850
Mean :0.02042 Mean :0.016069 Mean :0.013420 Mean :0.010709 Mean :0.010941 Mean :0.00929 Mean :0.008222
3rd Qu.:0.02527 3rd Qu.:0.020825 3rd Qu.:0.016725 3rd Qu.:0.014900 3rd Qu.:0.014500 3rd Qu.:0.01210 3rd Qu.:0.010575
Max. :0.08250 Max. :0.100400 Max. :0.070900 Max. :0.039000 Max. :0.035200 Max. :0.04470 Max. :0.039400
V57 V58 V59 V60 Class
Min. :0.00030 Min. :0.000300 Min. :0.000100 Min. :0.000600 M:111
1st Qu.:0.00370 1st Qu.:0.003600 1st Qu.:0.003675 1st Qu.:0.003100 R: 97
Median :0.00595 Median :0.005800 Median :0.006400 Median :0.005300
Mean :0.00782 Mean :0.007949 Mean :0.007941 Mean :0.006507
3rd Qu.:0.01043 3rd Qu.:0.010350 3rd Qu.:0.010325 3rd Qu.:0.008525
Max. :0.03550 Max. :0.044000 Max. :0.036400 Max. :0.043900 Setelah data dimuat, kita perlu membagi data menjadi data latih dan data uji. Data latih digunakan dalam proses pelatihan model serta mencari model terbaik, sementara data uji digunakan untuk mengevaluasi kemampuan model dalam memprediksi data baru yang belum pernah dilihat sebelumnya.
Untuk membagian data dapat menggunakan beberapa cara, salah satunya menggunakan fungsi createDataPartition dari package caret. Data yang akan digunakan sebagai data latih sebanyak 80 persen dan sisanya menjadi data uji.
R
# Membagi data latih dan data uji set.seed(123) indeks_latih <- createDataPartition(Sonar$Class, p = 0.8, list = FALSE) data_latih <- Sonar[indeks_latih, ] data_uji <- Sonar[-indeks_latih, ]
Model Dasar
Model pertama yang kita buat akan menggunakan fungsi train dan validasi menggunakan fungsi trainControl dengan metode "repeatedcv" (cross validation) dengan 10 fold dan 3 kali ulangan. Pada model pertama ini kita hanya akan menentukan parameter mtry (jumlah variabel yang digunakan pada setiap proses splitting) sebesar sqrt(n_var). Adapun evaluasi model akan menggunakan nilai accuracy.
R
control <- trainControl(method="repeatedcv", number=10, repeats = 3) n_var = ncol(data_latih) -1 mtry <- sqrt(n_var) # jumlah fitur untuk splitting node param_grid <- expand.grid(.mtry = mtry) rf_base <- train(Class~., data=data_latih, method="rf", metric="Accuracy", tuneGrid=param_grid, trControl=control) print(rf_base)
Output
# OUTPUT Random Forest 167 samples 60 predictor 2 classes: 'M', 'R' No pre-processing Resampling: Cross-Validated (10 fold, repeated 3 times) Summary of sample sizes: 150, 150, 150, 151, 150, 150, ... Resampling results: Accuracy Kappa 0.8159559 0.6253915 Tuning parameter 'mtry' was held constant at a value of 7.745967
Hasil yang diperoleh menunjukkan model ini memiliki akurasi sebesar 0.8159559. Nilai ini merupakan nilai rata-rata akurasi berdasarkan hasil repeated k-fold cv sebanyak 10 fold dengan 3 kali ulangan.
Tuning Hyperparameter (Random Search)
Model kedua yang akan dibuat adalah dengan mengatur parameter mtry melalui random search. Tahapan yang terjadi pada proses ini adalah pada proses pelatihan akan diambil sebanyak beberapa nilai acak untuk dijadikan parameter mtry. Banyaknya nilai acak ditentukan berdasarkan parameter tuneLength. Pada contoh ini kita menentukan nilai tuneLength=5.
R
# Random Search set.seed(1000) control <- trainControl(method="repeatedcv", number=10, repeats=3, search="random") mtry <- sqrt(ncol(data_latih)-1) rf_random <- train(Class~., data=data_latih, method="rf", metric="Accuracy", tuneLength=5, trControl=control) print(rf_random) plot(rf_random)
Output
# OUTPUT Random Forest 167 samples 60 predictor 2 classes: 'M', 'R' No pre-processing Resampling: Cross-Validated (10 fold, repeated 3 times) Summary of sample sizes: 150, 150, 150, 151, 150, 150, ... Resampling results across tuning parameters: mtry Accuracy Kappa 22 0.7997631 0.5928894 24 0.8060294 0.6050191 29 0.8039297 0.6011269 43 0.8017239 0.5968251 51 0.7859150 0.5639590 Accuracy was used to select the optimal model using the largest value. The final value used for the model was mtry = 24.
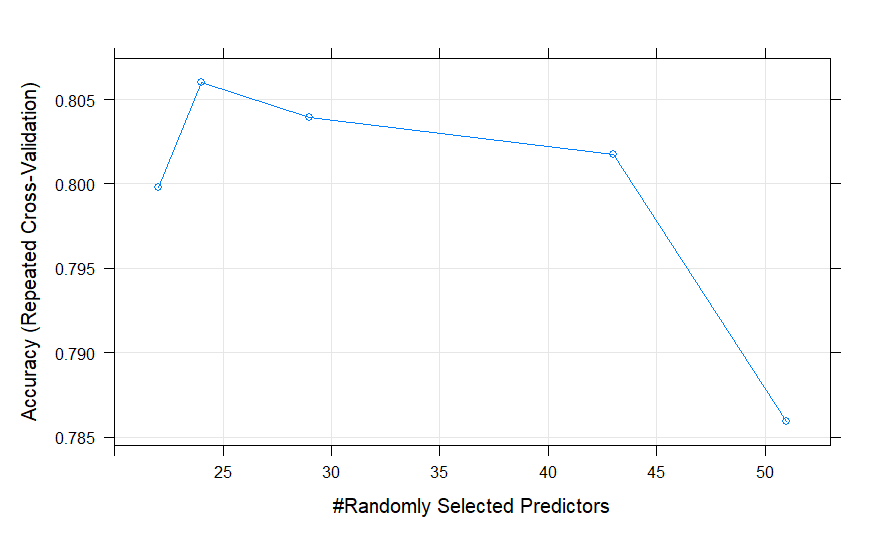

Pada model ini jumlah mtry yang dicobakan sebanyak 5 nilai yaitu 22, 24, 29, 43, 51. Berdasarkan kelima nilai mtry ini, diperoleh model dengan rata-rata nilai akurasi tertinggi pada data latih adalah mtry=24 yaitu sebesar 0.8060294. Hasil ini tidak sebaik sebelumnya, hal ini mungkin disebabkan karena kebetulan nilai mtry yang digunakan semuanya cukup besar, tidak ada nilai mtry kecil yang dicobakan dalam proses tuning hyperparameter-nya.
Tuning Hyperparameter (Grid Search)
Model ketiga akan dibangun menggunakan teknik grid search, teknik ini mirip dengan teknik random search hanya saja nilai mtry kita tentukan sendiri misalkan pada contoh ini sebanyak 5 nilai yaitu 1, 3, 5, 10 dan 15.
R
set.seed(1000) control <- trainControl(method="repeatedcv", number=10, repeats=3, search="grid") tunegrid <- expand.grid(.mtry=c(1,3,5,10,15)) rf_grid <- train(Class~., data=data_latih, method="rf", metric="Accuracy", tuneGrid=tunegrid, trControl=control) print(rf_grid) plot(rf_grid)
Output
# OUTPUT Random Forest 167 samples 60 predictor 2 classes: 'M', 'R' No pre-processing Resampling: Cross-Validated (10 fold, repeated 3 times) Summary of sample sizes: 150, 150, 150, 151, 150, 150, ... Resampling results across tuning parameters: mtry Accuracy Kappa 1 0.8175327 0.6281846 3 0.8137500 0.6204645 5 0.8305556 0.6559527 10 0.8159559 0.6253692 15 0.8081127 0.6089697 Accuracy was used to select the optimal model using the largest value. The final value used for the model was mtry = 5.


Dari output di atas diperoleh model dengan nilai mtry=5 memiliki rata-rata nilai Akurasi terbaik yaitu sebesar 0.8305556. Model ini juga memiliki nilai Akurasi yang lebih baik dibandingkan dua model sebelumnya.
Tuning Hyperparameter (Manual)
Tiga model yang sudah kita buat sebelumnya, hanya mengevaluasi model dengan mengatur nilai mtry saja, sementara parameter lainnya kita biarkan dengan nilai defaultnya. Fungsi randomForest sebenarnya memiliki beberapa parameter lain yang dapat kita atur untuk mencari model yang optimal. Beberapa diantaranya yaitu:
ntree: menunjukkan jumlah pohon yang ditumbuhkan dalam proses pelatihan modelnodesize: menentukan jumlah pengamatan minimum yang harus ada di setiap node terminal agar pemisahan lebih lanjut dilakukan saat membangun pohonmaxnodes: jumlah maksimum node terminal yang dapat dimiliki oleh setiap pohon.
Pada kode di bawah ini, kita membuat fungsi secara manual untuk melakukan tuning hiperparameter dengan teknik grid search. Pada fungsi ini kita dapat melakukan pencarian tidak hanya untuk parameter mtry tetapi juga ntree, nodesize dan maxnodes.
R
# Membuat fungsi untuk menangani pelatihan model dengan berbagai nilai parameter
rf_tune <- function(data, ntree, nodesize, maxnodes, tunegrid, control, seed=100) {
modellist <- list()
param_grid = expand.grid(ntree, nodesize, maxdepth)
names = c("ntree", "nodesize", "maxnodes")
param_grid <- setNames(param_grid, names)
for (i in 1:nrow(param_grid)){
nt = param_grid[i, "ntree"]
ns = param_grid[i, "nodesize"]
mn = param_grid[i, "maxnodes"]
set.seed(seed)
fit <- train(Class~., data=data, method="rf", metric="Accuracy",
tuneGrid=tunegrid, trControl=control,
ntree=nt, nodesize=ns, maxnodes=mn)
key <- toString(length(modellist)+1)
modellist[[key]] <- fit
}
return (modellist)
}
# Contoh penerapan
# Manual Grid Search
control <- trainControl(method="repeatedcv", number=10, repeats=3, search="grid", allowParallel = T)
tunegrid <- expand.grid(.mtry=c(3,5,7,10))
ntree_list <- c(100, 200, 500)
nodesize_list <- 1:5
maxnodes_list <- c(10,20,30,40)
all_model <- rf_tune(data_latih, ntree_list, nodesize_list, maxnodes_list, tunegrid, control, seed = 1000)
# compare results
results <- resamples(all_model)
summary(results)
dotplot(results)Output
Call:
summary.resamples(object = results)
Models: 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17,
18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34,
35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51,
52, 53, 54, 55, 56, 57, 58, 59, 60
Number of resamples: 30
Accuracy
Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
1 0.6250000 0.8125000 0.8235294 0.8296814 0.8823529 1 0
2 0.6470588 0.7647059 0.8235294 0.8364379 0.8823529 1 0
3 0.6470588 0.7647059 0.8235294 0.8284722 0.8823529 1 0
4 0.6250000 0.7647059 0.8235294 0.8196324 0.8823529 1 0
5 0.6250000 0.7647059 0.8235294 0.8114052 0.8805147 1 0
6 0.6470588 0.7766544 0.8235294 0.8300654 0.8823529 1 0
7 0.6000000 0.7647059 0.8235294 0.8153105 0.8729167 1 0
8 0.6470588 0.7647059 0.8235294 0.8296650 0.8823529 1 0
9 0.6470588 0.7766544 0.8235294 0.8298039 0.8823529 1 0
10 0.5882353 0.7647059 0.8235294 0.8241830 0.8823529 1 0
11 0.6470588 0.7647059 0.8235294 0.8203840 0.8823529 1 0
12 0.6470588 0.7647059 0.8235294 0.8220833 0.8729167 1 0
13 0.5882353 0.7647059 0.8235294 0.8159559 0.8823529 1 0
14 0.6875000 0.7647059 0.8235294 0.8279820 0.8823529 1 0
15 0.5882353 0.7647059 0.8235294 0.8141340 0.8750000 1 0
16 0.6250000 0.8125000 0.8235294 0.8296814 0.8823529 1 0
17 0.6470588 0.7647059 0.8235294 0.8364379 0.8823529 1 0
18 0.6470588 0.7647059 0.8235294 0.8284722 0.8823529 1 0
19 0.6250000 0.7647059 0.8235294 0.8196324 0.8823529 1 0
20 0.6250000 0.7647059 0.8235294 0.8114052 0.8805147 1 0
21 0.6470588 0.7766544 0.8235294 0.8300654 0.8823529 1 0
22 0.6000000 0.7647059 0.8235294 0.8153105 0.8729167 1 0
23 0.6470588 0.7647059 0.8235294 0.8296650 0.8823529 1 0
24 0.6470588 0.7766544 0.8235294 0.8298039 0.8823529 1 0
25 0.5882353 0.7647059 0.8235294 0.8241830 0.8823529 1 0
26 0.6470588 0.7647059 0.8235294 0.8203840 0.8823529 1 0
27 0.6470588 0.7647059 0.8235294 0.8220833 0.8729167 1 0
28 0.5882353 0.7647059 0.8235294 0.8159559 0.8823529 1 0
29 0.6875000 0.7647059 0.8235294 0.8279820 0.8823529 1 0
30 0.5882353 0.7647059 0.8235294 0.8141340 0.8750000 1 0
31 0.6250000 0.8125000 0.8235294 0.8296814 0.8823529 1 0
32 0.6470588 0.7647059 0.8235294 0.8364379 0.8823529 1 0
33 0.6470588 0.7647059 0.8235294 0.8284722 0.8823529 1 0
34 0.6250000 0.7647059 0.8235294 0.8196324 0.8823529 1 0
35 0.6250000 0.7647059 0.8235294 0.8114052 0.8805147 1 0
36 0.6470588 0.7766544 0.8235294 0.8300654 0.8823529 1 0
37 0.6000000 0.7647059 0.8235294 0.8153105 0.8729167 1 0
38 0.6470588 0.7647059 0.8235294 0.8296650 0.8823529 1 0
39 0.6470588 0.7766544 0.8235294 0.8298039 0.8823529 1 0
40 0.5882353 0.7647059 0.8235294 0.8241830 0.8823529 1 0
41 0.6470588 0.7647059 0.8235294 0.8203840 0.8823529 1 0
42 0.6470588 0.7647059 0.8235294 0.8220833 0.8729167 1 0
43 0.5882353 0.7647059 0.8235294 0.8159559 0.8823529 1 0
44 0.6875000 0.7647059 0.8235294 0.8279820 0.8823529 1 0
45 0.5882353 0.7647059 0.8235294 0.8141340 0.8750000 1 0
46 0.6250000 0.8125000 0.8235294 0.8296814 0.8823529 1 0
47 0.6470588 0.7647059 0.8235294 0.8364379 0.8823529 1 0
48 0.6470588 0.7647059 0.8235294 0.8284722 0.8823529 1 0
49 0.6250000 0.7647059 0.8235294 0.8196324 0.8823529 1 0
50 0.6250000 0.7647059 0.8235294 0.8114052 0.8805147 1 0
51 0.6470588 0.7766544 0.8235294 0.8300654 0.8823529 1 0
52 0.6000000 0.7647059 0.8235294 0.8153105 0.8729167 1 0
53 0.6470588 0.7647059 0.8235294 0.8296650 0.8823529 1 0
54 0.6470588 0.7766544 0.8235294 0.8298039 0.8823529 1 0
55 0.5882353 0.7647059 0.8235294 0.8241830 0.8823529 1 0
56 0.6470588 0.7647059 0.8235294 0.8203840 0.8823529 1 0
57 0.6470588 0.7647059 0.8235294 0.8220833 0.8729167 1 0
58 0.5882353 0.7647059 0.8235294 0.8159559 0.8823529 1 0
59 0.6875000 0.7647059 0.8235294 0.8279820 0.8823529 1 0
60 0.5882353 0.7647059 0.8235294 0.8141340 0.8750000 1 0

R
all_model[["47"]]$finalModel["param"]
Output
# OUTPUT $ntree [1] 200 $nodesize [1] 1 $maxnodes [1] 40
Dari hasil iterasi berbagai kombinasi nilai parameter di atas, diperoleh model dengan rata-rata akurasi tertinggi yaitu model 47. Model ini memiliki pengaturan mtry=3, ntree=200, nodesize=1 dan maxnodes=40.
Evaluasi Model
Langkah selanjutnya yaitu melakukan evaluasi model yang telah dibangun menggunakan data uji. Data uji yang digunakan merupakan data yang sama sekali belum pernah dilihat oleh model, sehingga prediksi yang dihasilkan dapat mencerminkan dengan baik kemampuan model yang sesungguhnya.
R
pred_base <- predict(rf_base, data_uji)
pred_rs <- predict(rf_random, data_uji)
pred_gs <-predict(rf_grid, data_uji)
pred_man <- predict(all_model["47"], data_uji)
confusionMatrix(pred_base, reference = data_uji$Class)
print("============================================")
confusionMatrix(pred_rs, reference = data_uji$Class)
print("============================================")
confusionMatrix(pred_gs, reference = data_uji$Class)
print("============================================")
confusionMatrix(pred_man$"47", reference = data_uji$Class)Output
# OUTPUT
Confusion Matrix and Statistics
Reference
Prediction M R
M 19 0
R 3 19
Accuracy : 0.9268
95% CI : (0.8008, 0.9846)
No Information Rate : 0.5366
P-Value [Acc > NIR] : 6.182e-08
Kappa : 0.8544
Mcnemar's Test P-Value : 0.2482
Sensitivity : 0.8636
Specificity : 1.0000
Pos Pred Value : 1.0000
Neg Pred Value : 0.8636
Prevalence : 0.5366
Detection Rate : 0.4634
Detection Prevalence : 0.4634
Balanced Accuracy : 0.9318
'Positive' Class : M
============================================
Confusion Matrix and Statistics
Reference
Prediction M R
M 18 1
R 4 18
Accuracy : 0.878
95% CI : (0.738, 0.9592)
No Information Rate : 0.5366
P-Value [Acc > NIR] : 3.487e-06
Kappa : 0.7574
Mcnemar's Test P-Value : 0.3711
Sensitivity : 0.8182
Specificity : 0.9474
Pos Pred Value : 0.9474
Neg Pred Value : 0.8182
Prevalence : 0.5366
Detection Rate : 0.4390
Detection Prevalence : 0.4634
Balanced Accuracy : 0.8828
'Positive' Class : M
============================================
Confusion Matrix and Statistics
Reference
Prediction M R
M 20 0
R 2 19
Accuracy : 0.9512
95% CI : (0.8347, 0.994)
No Information Rate : 0.5366
P-Value [Acc > NIR] : 5.331e-09
Kappa : 0.9026
Mcnemar's Test P-Value : 0.4795
Sensitivity : 0.9091
Specificity : 1.0000
Pos Pred Value : 1.0000
Neg Pred Value : 0.9048
Prevalence : 0.5366
Detection Rate : 0.4878
Detection Prevalence : 0.4878
Balanced Accuracy : 0.9545
'Positive' Class : M
============================================
Confusion Matrix and Statistics
Reference
Prediction M R
M 21 0
R 1 19
Accuracy : 0.9756
95% CI : (0.8714, 0.9994)
No Information Rate : 0.5366
P-Value [Acc > NIR] : 2.995e-10
Kappa : 0.9511
Mcnemar's Test P-Value : 1
Sensitivity : 0.9545
Specificity : 1.0000
Pos Pred Value : 1.0000
Neg Pred Value : 0.9500
Prevalence : 0.5366
Detection Rate : 0.5122
Detection Prevalence : 0.5122
Balanced Accuracy : 0.9773
'Positive' Class : M Dari output di atas dapat dilihat bahwa model terakhir yaitu model terbaik hasil tuning secara manual memiliki nilai akurasi paling tinggi yaitu sebesar 0.9756, dimana model hanya salah memprediksi 1 amatan yang seharusnya masuk kelas M namun diprediksi sebagai kelas R, Adapun model lainnya memiliki nilai akurasi yang bervariasi yaitu 0.9268, 0.878 dan 0.9512. Nilai-nilai yang diperoleh pada masing-masing model pada contoh ini kebetulan sangat tinggi bahkan jauh lebih tinggi dibandingkan rata-rata hasil evaluasi pada data latih.
Bonus : doParallel
Proses tuning hyperparameter pada model randomforest memerlukan waktu yang cukup lama khususnya ketika data semakin banyak dan parameter yang dituning juga lebih bervariasi. Secara default kompiler R hanya menggunakan 1 core CPU saja dalam mengeksekusi kode. Kita dapat menggunakan jumlah core yang lebih banyak dengan menggunakan fitur pada package doParallel. Kita juga perlu memastikan parameter allowParallel pada fungsi trainControl bernilai TRUE.
R
library(doParallel)
start_time_paralel <- system.time({
# Menggunakan 3 inti CPU
cl <- makeCluster(3)
# Mengaktifkan cluster
registerDoParallel(cl)
# Manual Search
control <- trainControl(method="repeatedcv", number=10, repeats=3, search="grid", allowParallel = T)
tunegrid <- expand.grid(.mtry=c(3,5,7,10))
ntree_list <- c(100, 200, 500)
nodesize_list <- 1:5
maxnodes_list <- c(10,20,30,40)
all_model <- rf_tune(data_latih, ntree_list, nodesize_list, maxnodes_list, tunegrid, control, seed = 1000)
# Menghentikan cluster
stopCluster(cl)
})[["elapsed"]]
print(paste("Waktu eksekusi multicore:", start_time_paralel, "detik"))
# =================================================================#
# Eksekusi kode yang sama tanpa paralel
start_time_normal <- system.time({
# Manual Search
control <- trainControl(method="repeatedcv", number=10, repeats=3, search="grid")
tunegrid <- expand.grid(.mtry=c(3,5,7,10))
ntree_list <- c(100, 200, 500)
nodesize_list <- 1:5
maxnodes_list <- c(10,20,30,40)
all_model <- rf_tune(data_latih, ntree_list, nodesize_list, maxnodes_list, tunegrid, control, seed = 1000)
})[["elapsed"]]
print(paste("Waktu eksekusi normal:", start_time_normal, "detik"))
Output
# OUTPUT [1] "Waktu eksekusi multicore: 310.849999999999 detik" [1] "Waktu eksekusi normal: 381.800000000003 detik"
Dari hasil tersebut terdapat perbedaan waktu eksekusi yang signifikan antara penggunaan doParallel dan tidak. Hasil ini tentu sangat tergantung juga dengan berbagai faktor, baik hardware dan sofware. Namun pada lingkungan pemrosesan yang sama, penggunaan lebih banyak core tentu akan memberikan waktu yang lebih cepat.
Perlu menjadi catatan, jika melakukan proses secara paralel, akan menghasilkan model yang berbeda bahkan ketika menggunakan nilai set.seed yang sama. Jika ingin benar-benar mereproduksi output yang sama persis, maka kita perlu menetapkan parameter seeds pada fungsi trainControl (misalkan sebanyak 10 x 3 + 1= 31 nilai seed) . Jumlah ini yaitu jumlah number dikali nilai repeats pada repeatedcv (lihat trainControl Docs).







maaf kak, mau nanya. untuk random search, apakah bisa dilakukan tuning selain di parameter mtry?
Bisa kak, coba cek di tulisan ini https://sainsdata.id/machine-learning/8544/model-machine-learning-dengan-tidymodels-r/, nanti silahkan lihat dibagian random forest dan tuningnya dengan tidymodels