Tuning Hyperparameter Model Random Forest dengan Bayesian Optimization di R


Tuning hyperparameter adalah bagian penting dalam pengembangan model machine learning, termasuk pada Random Forest. Pemilihan hyperparameter yang optimal mampu meningkatkan performa model secara signifikan, sementara pengaturan yang kurang tepat mungkin dapat menurunkan akurasi prediksi model. Terdapat beberapa pendekatan yang sering digunakan, mulai dari Grid Search, Random Search, hingga Bayesian Optimization.
Bayesian Optimization sendiri merupakan metode pencarian hyperparameter berbasis probabilistik yang efisien dalam menemukan kombinasi parameter terbaik. Teknik ini memodelkan fungsi objektif dengan distribusi tertentu, lalu memanfaatkan model tersebut untuk mengarahkan pencarian ke area hyperparameter yang menjanjikan. Dengan cara ini, Bayesian Optimization dapat melakukan eksplorasi dan eksploitasi secara efektif sehingga mampu menemukan hyperparameter optimal dengan lebih sedikit iterasi dibanding teknik grid search maupun random search.
lihat: Bayesian Optimization: Teori dan Intuisi
Pada tutorial ini, akan dijelaskan secara step by step bagaimana melakukan tuning hyperparameter model Random Forest di R menggunakan Bayesian Optimization. Library utama yang digunakan adalah ParBayesianOptimization untuk optimasi dan ranger untuk model Random Forest.
Persiapan Data dan Library
Pada contoh ini, dataset yang digunakan adalah data wine quality binary yang dapat diakses langsung pada tautan berikut: https://raw.githubusercontent.com/sainsdataid/dataset/main/wine-quality-binary.csv.
R
# Instalasi package jika belum tersedia
# install.packages(c("ranger", "ParBayesianOptimization", "caret"))
# Load library yang dibutuhkan
library(ranger) # Random Forest
library(ParBayesianOptimization) # Bayesian Optimization
library(caret) # Untuk evaluasi dan split data
# Memuat dataset dari URL
data_wine <- read.csv("https://raw.githubusercontent.com/sainsdataid/dataset/main/wine-quality-binary.csv")
# Melihat struktur data
str(data_wine)# OUTPUT 'data.frame': 1143 obs. of 13 variables: $ id : int 1 2 3 4 5 6 7 8 9 10 ... $ fixed.acidity : num 7.4 7.8 7.8 11.2 7.4 7.4 7.9 7.3 7.8 6.7 ... $ volatile.acidity : num 0.7 0.88 0.76 0.28 0.7 0.66 0.6 0.65 0.58 0.58 ... $ citric.acid : num 0 0 0.04 0.56 0 0 0.06 0 0.02 0.08 ... $ residual.sugar : num 1.9 2.6 2.3 1.9 1.9 1.8 1.6 1.2 2 1.8 ... $ chlorides : num 0.076 0.098 0.092 0.075 0.076 0.075 0.069 0.065 0.073 0.097 ... $ free.sulfur.dioxide : num 11 25 15 17 11 13 15 15 9 15 ... $ total.sulfur.dioxide: num 34 67 54 60 34 40 59 21 18 65 ... $ density : num 0.998 0.997 0.997 0.998 0.998 ... $ pH : num 3.51 3.2 3.26 3.16 3.51 3.51 3.3 3.39 3.36 3.28 ... $ sulphates : num 0.56 0.68 0.65 0.58 0.56 0.56 0.46 0.47 0.57 0.54 ... $ alcohol : num 9.4 9.8 9.8 9.8 9.4 9.4 9.4 10 9.5 9.2 ... $ quality : chr "LOW" "LOW" "LOW" "HIGH" ...
Preprocessing Data
Dalam pemodelan, sangat disarankan untuk melakukan eksplorasi data secara mendalam agar memahami karakteristik data dengan lebih baik. Namun pada tulisan ini kita akan fokus pada tahapan pemodelan dan tuning hyperparameter menggunakan Bayesian Optimization. Silahkan pembaca melakukan eksplorasi secara mandiri jika diinginkan.
Pada tahap ini, kita perlu memastikan bahwa data sudah siap digunakan untuk modeling. Kolom target pada dataset ini adalah quality, yang bertipe teks ("LOW" dan dan "HIGH"). Agar kompatibel dengan model klasifikasi, maka ubah menjadi tipe faktor. Kemudian, pada bagian ini juga kita lakukan pembagian data menjadidata latih dan data uji menggunakan fungsi createDataPartition dari paket caret.
R
set.seed(123) # Ubah kolom target menjadi faktor data_wine$quality <- as.factor(data_wine$quality) # Split data 80% train, 20% test data_split <- createDataPartition(data_wine$quality, p = 0.8, list = FALSE) data_train <- data_wine[data_split, ] data_test <- data_wine[-data_split, ] # Cek proporsi data table(data_train$quality) table(data_test$quality)
# OUTPUT HIGH LOW 497 418 HIGH LOW 124 104
Membuat Fungsi Evaluasi
Sebelum melakukan pencarian hyperparameter, kita perlu mendefinisikan sebuah fungsi evaluasi yang akan digunakan nantinya dalam proses Bayesian Optimization. Fungsi ini akan mengambil nilai-nilai hyperparameter sebagai input, melatih model Random Forest dengan parameter tersebut (menggunakan cross-validation), lalu mengembalikan metrik kinerja model (misal akurasi rata-rata) sebagai skor.
Berikut ini merupakan beberapa hyperparameter penting untuk tuning model random forest dari paket ranger:
num.trees: jumlah pohonmtry: jumlah variabel yang dipilih pada setiap splitmin.node.size: ukuran minimal sampel pada node terminalmax.depth: kedalaman maksimal pohonsample.fraction: proporsi sampel yang diambil pada setiap pohon- selengkapnya dapat dilihat pada dokumentasi
ranger(docs)
Fungsi evaluasi berikut menggunakan cross-validation 5-fold dan mengembalikan akurasi rata-rata sebagai skor kebaikan model.
R
rf_cv_bayes <- function(num.trees, mtry, min.node.size, max.depth, sample.fraction) {
num.trees <- as.integer(num.trees)
mtry <- as.integer(mtry)
min.node.size <- as.integer(min.node.size)
max.depth <- as.integer(max.depth)
sample.fraction <- as.numeric(sample.fraction)
set.seed(123)
ctrl <- trainControl(method = "cv", number = 5)
model <- train(quality ~ ., data = data_train,
method = "ranger",
metric = "Accuracy",
trControl = ctrl,
tuneGrid = data.frame(
mtry = mtry,
splitrule = "gini",
min.node.size = min.node.size
),
num.trees = num.trees,
max.depth = max.depth,
sample.fraction = sample.fraction)
list(Score = getTrainPerf(model)$TrainAccuracy)
}Menentukan Ruang Pencarian Hyperparameter
Ruang pencarian (search space) adalah rentang nilai yang akan dieksplorasi oleh Bayesian Optimization untuk setiap hyperparameter. Semakin luas ruang pencarian, semakin besar kemungkinan menemukan kombinasi terbaik, namun juga memerlukan waktu komputasi yang lebih lama.
Pada contoh ini, ruang pencarian dibuat sebagai berikut:
num.trees: Mulai dari 100 hingga 300 pohon. Biasanya nilai lebih besar akan memberikan model lebih stabil, namun terlalu banyak pohon juga memperlambat prosesmtry: Mulai dari 1 sampai jumlah fitur pada data, dikurangi 1 (karena kolom target tidak dihitung)min.node.size: Minimum sampel pada node terminal, 1 sampai 10max.depth: Kedalaman maksimum pohon, diatur dari 1 hingga 20
R
bounds <- list( num.trees = c(100L, 500L), mtry = c(1L, ncol(data_train)-1L), min.node.size = c(1L, 10L), max.depth = c(1L, 20L), sample.fraction = c(0.5, 1.0) )
Pencarian dengan Bayesian Optimization
Setelah fungsi evaluasi dan ruang pencarian dibuat, proses optimasi hyperparameter dapat dijalankan dengan Bayesian Optimization melalui fungsi bayesOpt.
Adapun penjelasan parameter pada fungsi bayesOpt yaitu:
initPoints: Jumlah titik awal yang dipilih secara acak sebelum algoritma mulai melakukan pencarian berbasis modeliters.n: Jumlah iterasi yang dilakukan setelah fase awalacq: Tipe fungsi akuisisi, di sini"ei"(Expected Improvement), yaitu memilih titik yang paling menjanjikan untuk perbaikan skor, fungsi lainnya yaitu"ucb"untuk Upper Confidence Bound, “poi” Probability of Improvement, dan"eips"untuk Expected Improvement Per Second.verbose: Apakah proses akan menampilkan log setiap iterasi (1 berarti menampilkan log untuk setiap iterasi)
Saat menjalankan fungsi ini maka Bayesian Optimization akan secara otomatis:
- Memilih kombinasi awal secara acak (
initPoints) untuk eksplorasi - Menggunakan model probabilistik (Gaussian Process) untuk memodelkan hubungan hyperparameter dengan hasil evaluasi
- Memilih kombinasi baru berdasarkan Expected Improvement (
acq = "ei") secara adaptif - Mencatat seluruh kombinasi dan skor selama iterasi
Setelah proses selesai, seluruh riwayat pencarian dan hasil evaluasi dapat dilihat dan digunakan untuk menemukan kombinasi hyperparameter terbaik sesuai batas-batas yang diberikan.
R
set.seed(123) # Pencarian dengan Bayesian Optimization opt_obj <- bayesOpt( FUN = rf_cv_bayes, bounds = bounds, initPoints = 10, iters.n = 20, acq = "ei", verbose = 1 )
# OUTPUT Running initial scoring function 10 times in 1 thread(s)... 10.15 seconds Starting Epoch 1 1) Fitting Gaussian Process... 2) Running local optimum search... 16.12 seconds 3) Running FUN 1 times in 1 thread(s)... 0.8 seconds Starting Epoch 2 1) Fitting Gaussian Process... 2) Running local optimum search... 15.84 seconds 3) Running FUN 1 times in 1 thread(s)... 0.65 seconds .... Starting Epoch 20 1) Fitting Gaussian Process... 2) Running local optimum search... 31.2 seconds 3) Running FUN 1 times in 1 thread(s)... 0.63 seconds
Jika diperlukan, objek opt_obj juga dapat divisualisasikan melalui fungsi plot. Grafik yang ditampilkan menunjukkan perkembangan nilai skor (misalnya akurasi) dari setiap iterasi pencarian hyperparameter selama proses Bayesian Optimization.
R
plot(opt_obj)
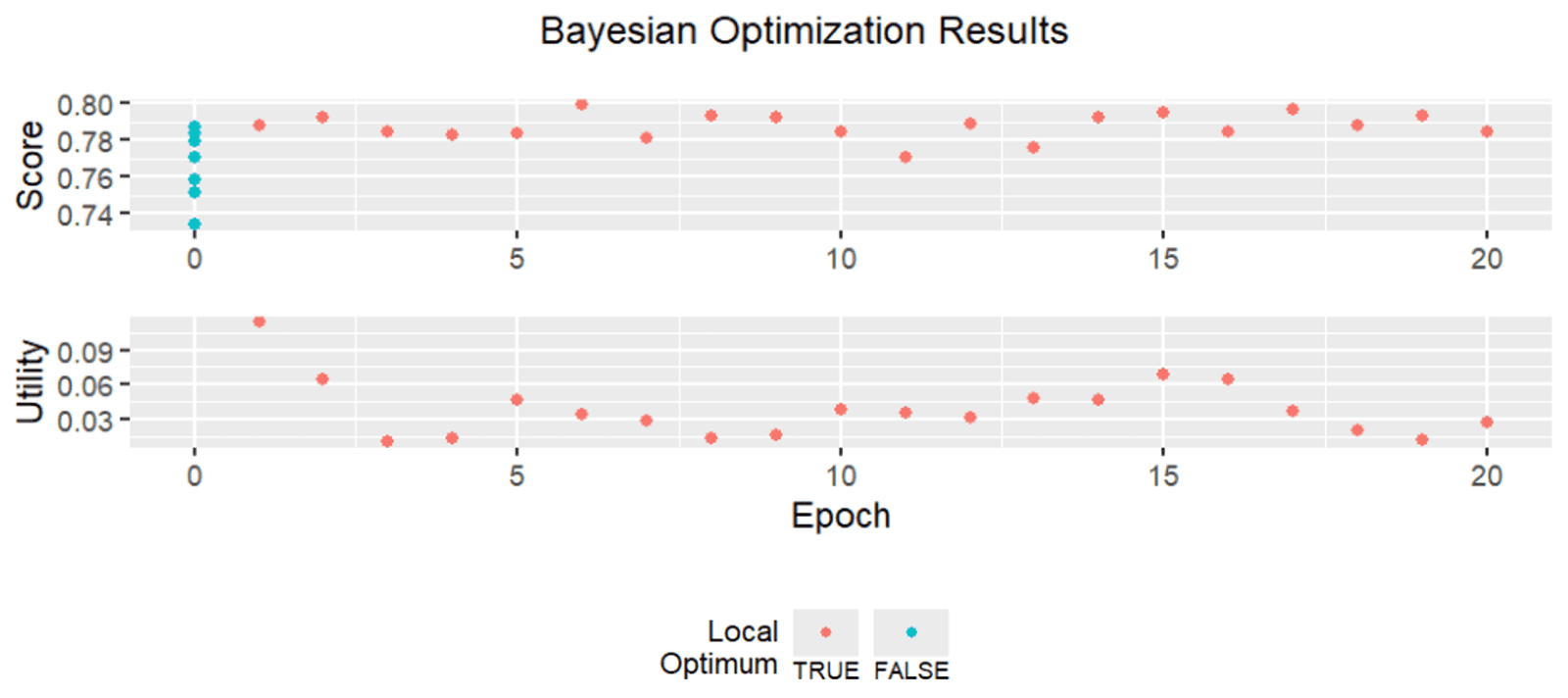

Membuat Model Terbaik
Setelah proses pencarian selesai, kita dapat mengambil kombinasi hyperparameter terbaik (dengan skor tertinggi dari seluruh iterasi yang telah dicoba). Nilai-nilai hyperparameter ini dapat diperoleh melalui fungsi getBestPars.
Kemudian, hyperparameter yang diperoleh dari proses tuning inilah yang akan digunakan untuk melatih model Random Forest akhir pada seluruh data latih. Dengan demikian, model final ini diharapkan memiliki performa terbaik berdasarkan proses pencarian yang telah dilakukan.
R
# Mengambil kombinasi hyperparameter terbaik dari hasil tuning tuned_params <- getBestPars(opt_obj) print(tuned_params) # Melatih model random forest dengan hyperparameter optimal model_final <- ranger( formula = quality ~ ., data = data_train, num.trees = as.integer(tuned_params$num.trees), mtry = as.integer(tuned_params$mtry), min.node.size = as.integer(tuned_params$min.node.size), max.depth = as.integer(tuned_params$max.depth), sample.fraction = as.numeric(tuned_params$sample.fraction), importance = "impurity", # Menyimpan informasi importance variabel classification = TRUE # Untuk klasifikasi biner )
# OUTPUT $num.trees [1] 100 $mtry [1] 4 $min.node.size [1] 4 $max.depth [1] 14 $sample.fraction [1] 0.9258608
Evaluasi Model
Setelah model akhir terbentuk menggunakan data latih dan hyperparameter hasil tuning, langkah selanjutnya adalah melakukan prediksi pada data uji yang belum pernah dilihat model.
R
# Prediksi pada data uji prediksi <- predict(model_final, data_test)$predictions # Confusion Matrix tabel_conf <- confusionMatrix(prediksi, data_test$quality) print(tabel_conf)
# OUTPUT
Confusion Matrix and Statistics
Reference
Prediction HIGH LOW
HIGH 100 22
LOW 24 82
Accuracy : 0.7982
95% CI : (0.7402, 0.8483)
No Information Rate : 0.5439
P-Value [Acc > NIR] : 8.644e-16
Kappa : 0.594
Mcnemar's Test P-Value : 0.8828
Sensitivity : 0.8065
Specificity : 0.7885
Pos Pred Value : 0.8197
Neg Pred Value : 0.7736
Prevalence : 0.5439
Detection Rate : 0.4386
Detection Prevalence : 0.5351
Balanced Accuracy : 0.7975
'Positive' Class : HIGH Kesimpulan
Bayesian Optimization mempermudah tuning hyperparameter model Random Forest di R secara efisien, khususnya untuk kombinasi parameter yang kompleks dan multidimensi. Dengan pendekatan ini, proses tuning menjadi lebih sistematis dan hasil yang diperoleh umumnya lebih baik dibandingkan teknik seperti grid search atau random search.
Referensi
- https://cran.r-project.org/web/packages/ParBayesianOptimization
- https://cran.r-project.org/web/packages/ranger
- https://topepo.github.io/caret/